
今天小编给大家介绍一款由上海交通大学与中国教育部系统控制与信息处理重点实验室共同研发的学习框架-ZeroBind学习框架,主要用于从蛋白质和药物的结构中预测蛋白质-药物相互作用;同时它还引入了弱监督的亚图信息量以及压缩的亚图作为潜在的结合口袋。ZeroBind通过任务自适应自注意模块训练了多个蛋白质的模型,自动学习了它们的重要性来进行最终预测,在药物靶标相互作用预测方面表现出了优越的性能。特别是对于那些未被见过的蛋白质和药物。下面我们来看下ZeroBind是如何预测蛋白质和药物互作的!

题目:ZeroBind: a protein-specific zero-shot predictor with subgraph matching for drugtarget interactions
译名:ZeroBind:基于亚图匹配的蛋白特异性零样本预测器,适用于药物靶点相互作用
期刊:Nature Communications
影响因子:IF:16.6
发表时间:2023.11.29
通讯作者:潘晓勇
文章亮点
ZeroBind方法将未知药物与蛋白的相互作用预测问题化为零样本学习问题,通过在现有蛋白质、药物和它们的相互作用上进行元学习,提高了对未知蛋白和药物的预测能力。
ZeroBind方法为每个蛋白质训练一个DTI任务模型,其中设计了任务自适应自注意力来计算多个DT任务对特定蛋白质元学习者的贡献;
ZeroBind方法提出了IB-子图学习方法,自动发现蛋白质中潜在的结合口袋,而不是派生出整个蛋白质的冗余图信息
ZeroBind方法在三个独立的零样本测试集和一个少样本测试集上进行了大量实验,数据结果表明ZeroBind始终优于现有方法。
ZeroBind方法介绍

图1 ZeBind学习框架。
包括四个主要模块:
图卷积网络编码器用于学习分子图和蛋白图的嵌入;
子图信息瓶颈模块用于生成蛋白图的重要信息子图作为潜在的结合口袋;
多层感知器模块用于连接蛋白质IB子图嵌入和分子嵌入以进行DTI预测;(4) 任务自适应自注意力模块用于度量不同任务的重要性。生成的任务权重用于加权平均损失,并进一步纳入元学习过程。
研究方法
作者们对数据集Generation andaugmentation进行了操作,进而建立高质量的基准数据集。首先,他们从BindingDB数据库中获取了超过260万个蛋白质与药物之间的结合亲和力数据。然后对这些数据进行了多个筛选和预处理步骤,如筛选“single protein”和特定类型的动力学常数。此外,还对目标蛋白进行了过滤,使其为人类或类人类蛋白。
为了展示ZeroBind的有效性,作者们构建了三个独立的测试集来评估模型性能。首先将蛋白质进行了序列相似性聚类,并通过分子骨架分割将分子分为训练和测试集。然后,构建了训练集、Transductive测试集、Semi-inductive测试集、Inductive测试集,用于在模型训练和评估过程中使用。另外,还利用网络负采样方法对训练数据集进行数据增强,以减轻注释不平衡问题。此外,作者们构建了一个二部图的药物靶蛋白网络,使用Djkstra算法找到网络中任意两个节点之间的最短路径距离,从而确定非结合对。

图 2 ZeroBind能够以弱监督的方式检测蛋白质的结合口袋。
作者们下载了996个蛋白质的3D结构并利用AlphaFold2对其余的635个蛋白质进行了结构预测。同时从PDBbind数据库中获取了真实的结合口袋信息,用于蛋白质和药物的图构建。
在图构建方面,作者描述了药物图和蛋白质图的构建过程。首先使用RDkit来构建分子,利用OGB数据集的编码格式来获取分子表示。蛋白质图的节点特征和边特征分别根据3D结构和预训练的ESM-2 embeddings进行初始化。整个过程包括从3D结构中构建蛋白质图和分子图。其次,在ZeroBind中使用元学习框架来训练蛋白质特异性模型,并定义为DTI任务、zero-shot DTI预测任务和few-shot DTI预测任务,展示了相应的预测流程。最后,作者应用了多种技术和改进方法来提高ZeroBind的性能,包括多步损失优化、学习每层每步的学习率和梯度方向、子图信息瓶颈和任务自适应自注意力等。通过以上操作,作者对ZeroBind进行了充分的数据处理和模型准备,为进一步的模型训练和推理做好了准备。
模型训练
在ZeroBind的训练过程中,模型首先使用任务的支持集更新到特定任务的模型,然后计算该任务的查询集的损失。重复N个内部步骤后,所有查询集的损失被加权平均,并用于通过梯度下降来优化元模型。ZeroBind的基本模型结构包括GNN模块、SIB模块和密集连接层,用于获得分子和蛋白质的嵌入表示并评估相互作用。具体地,ZeroBind采图神经网络(GNN)对分子和蛋白质的图进行表示学习,同时采用信息瓶颈原理(IB) 来识别潜在的蛋白质结合口袋的亚图。在SIB模块中通过模型无关的方法,识别出与DTI任务相关的关键信息,并排除嘈杂和多余的图信息。
ZeroBind模型与其他模型的比较
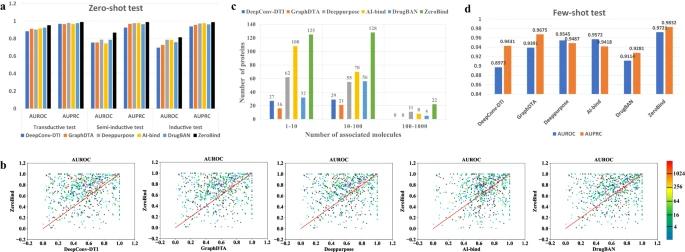
图 3 ZeroBind在零样本和少样本情境下与基准方法的性能比较
为了证明ZeroBind学习框架优于其他模型,作者们将ZeroBind与DeepConv-DTI、GraphDTA、 Deeppurpose、 Al-bind和DrugBAN这五种模型的优势进行比较评估,并在三个独立的测试集和一个小样本测试集上计算了接收器操作特性曲线下面积(AUROC) 和精确率回率曲线下面积(AUPRC)。结果表明,ZeroBind学习的模型能够很好地预测未见分子和蛋白质的DTI,也能在少量训练样本下迅速适应额外的蛋白质任务。这是一个涉及到元学习和深度神经网络的高级技术内容,需要读者具有一定的背景知识。
研究数据来源
作者们的研究成果是基于在线网络服务器和多个数据库的基准数据集和实验蛋白质结构数据获取的,并且这些数据可以通过以下网站免费获取:
在线网络服务器:http://www.csbio.sjtu.edu.cn/bioinf/ZeroBind/
基准数据集来源: BindingDB(https://www.bindingdb.org/bind/downloads/BindingDB_ All_ 2D_ 202311 sdf.zip)
SARS-COV-2测试数据集和实验蛋白质结构数据:
http://www.csbio.sjtu.edu.cn/bioinf/ZeroBind/datasets.html RCSB PDB
数据库:https://www.rcsb.org/downloads/
AlphaFold蛋白质结构数据库
https://www.alphafold.ebi.ac.uk/
PDB和AlphaFold代码
https://github.com/myprecioushh/ZeroBind
https://github.com/myprecioushh/ZeroBind
文章小结
ZeroBind是一个预训练模型,在少样本微调集上进行微调,并在相应的少样本测试集上进行评估,结果相对提高了AUROC 1.55%和AUPRC1.62%,优于最佳基线方法。
2、Meta-learning框架具有强大的泛化能力,能够快速适应具有少量训练样本的附加蛋白质任务。它在弱监督方式下检测与已知蛋白质结合口袋对齐良好的子图,并能够预测潜在药物,验证了其预测的可靠性。
3、通过模块删除实验发现去除Meta-learning策略、 SIB模块、任务适应注意力模块或使用其他GCN代替GCN都会降低模型性能,表明这些模块对模型的有效性具有重要作用。
